Découverte de la bioinformatique
(stage de collège)
Introduction
Qu'est ce que la bioinformatique ? Comme vous pouvez l'entendre c'est le mélange de la bio (biologie) et de l' informatique, ou plutôt de l'informatique spécialisé pour la biologie. L'informatique est maintenant partout, à la maison avec la domotique, dans les banques ou encore dans votre télé. Dans tous les cas il y a de l'informatique mais avec des programmes spécialisés écrits par des gens spécialistes de leur domaine. En bioinformatique on les appelle bionformaticien. Un bioinformaticien a besoin d'avoir des bases en génétique/biologie pour développer des programmes spécifiques. Il faut une bonne compréhension du fonctionnement d'un être vivant, de sa composition et donc de tout de qui tourne autour de l'ADN et des gènes. Qu'est ce que l'ADN ? les gènes ?
Une petite vidéo explicative sur diverses notions:
Une vidéo sur les notions de séquences d'ADN et de bioinformatique:
Maintenant que les bases de l'ADN et de la notion de séquence d'ADN ont été vues, nous allons réaliser ensemble un petit TP de phylogénie moléculaire. C'est à dire que nous allons récupérer des séquences de génome de différents organismes et grâce à ces séquences nous allons construire une sorte d'arbre. Cet arbre montrera la distance évolutive entre les organismes, c'est à dire "la ressemblance" plus ou moins important des espèces entre elles. On utilise l'image de l'arbre pour montrer l'évolution des espèces les unes par rapport aux autres car il permet de pouvoir positionner chaque espèce comme une feuille d'un arbre et de définir des branches plus ou moins longues selon les ressemblances entre les espèces.
Quelles sont les étapes que nous allons réaliser ?
- 1) Explorer des banques de données pour récupérer les séquences de génome de différents organismes
- 2) Comparer ces séquences pour extraire les informations identiques et différentes
- 3) Utiliser les informations de comparaison pour construire un arbre qui transformera les différences de séquences en branches de différentes longueurs
Les grandes banques de données
Comme vous avez pu le lire, un des aspects de la bioinformatique concerne la creation de grandes banques de données. Cela permet de rendre les données scientifique facilement accessible à tout le monde et pouvoir ainsi centraliser les informations. Pour que cela soit possible, il faut pouvoir disposer de grands centres de stockage de la même façon que vous utilisez des centres de stockage ("datacenter", "cloud") pour stocker les photos ou la musique de votre téléphone. Les données biologiques peuvent générer des fichiers très gros, ce qui nécessite beaucoup de place et donc beaucoup d'argent. La gestion de ces grandes banques de données est donc assurée par de grands centres de recherche internationaux qui disposent de très grosses machines.
Il existe un institut incontournable pour les données biologiques: NCBI (National Center for Biotechnology Information) qui est originellement un institut de medecine américain.
Le NCBI abrite la plus grande banque de données internationale concernant les informations genomiques, à savoir les séquences des génomes, les données d'expression des gènes, les protéines, ... Pour explorer cette grande banque de données: ici
Tapez n'importe quel mot (en anglais) et lancer la recherche. Nous allons regarder ensemble les résultats.
L'alignement de séquence
Nous avons vu qu'il est possible de récupérer facilement des séquences de n'importe quel organisme à partir de mots-clés dans la banque du NCBI.Avec ces séquences nous pouvons réaliser de nombreuses analyses. Une des choses les plus courantes en bioinformatique consiste à regarder la ressemblance des séquences entre elle en réalisant ce que l'on appelle de l'alignement de séquences. Pour cela nous allons utiliser un logiciel qui va permettre de pouvoir mettre en évidence les ressemblances et les différences.
Je vous propose d'aligner des séquences d'ARN 28S d'Homme, de gorille, d'orang-outan, de rat, de souris et de dingo. Pour information, l'ARN 28S est un élément essentiel pour la construction de ce que l'on appelle les ribosomes. Les ribosomes sont les "usines" à protéines, ce sont eux qui vont permettre de transformer l'expression d'un gène (ARNm) en protéine. Les ribosomes sont donc des éléments très importants et conservés. Ainsi il y a de forte chances que la séquence d'un ARN 28S d'Homme soit proche de celle d'un Gorille.
Récuperez le fichier sequences.fasta: ici, qui contient les séquences qui nous intéressent. Vous pouvez regarder le fichier avec un éditeur de texte.
Ouvrez la page du logiciel MultAlin et chargez le fichier select a file: Parcourir. Puis lancez le logiciel "Start MultAlin"
Nous allons regarder ensemble les résultats.
Les arbres phylogénétiques
Nous savons maintenant aligner des séquences de différents organismes et mettre en évidences les parties communes et différentes entre ces séquences. A partir de ces différences nous allons essayer de mettre en évidence la distance évolutive entre ces organismes. C'est à dire que nous allons essayer de déduire quels sont les organismes/espèces les plus proches sur l'arbre des espèces. En effet, si on suppose que des organismes ou espèces sont proches, il y a de grandes chances que leurs séquences soient plus proches en comparaison avec des espèces éloignées. Par exemple, dans l'exemple que nous étudions, nous pouvons imaginer que la souris est plus proche du rat que de l'Homme. De même on suppose que le Gorille est plus proche de l'Homme que la souris. Pour vérifier cette hypothèse nous allons réaliser un arbre phylogénétique. Grâce à des logiciels qui utilisent les différences entre les séquences nous allons pouvoir établir ce que l'on nomme des distances. Ces distances seront calculées pour toutes les combinaisons d'espèces et nous permettrons de créér des liens plus ou moins long entre chaque espèce. Pour réaliser cette analyse nous allons utiliser le site phylogenie.fr.
Choisissez Phylogeny Analysis dans le menu, puis One Click. Selectionnez votre fichier sequences.fasta puis lancer l'analyse Submit.
Vous allez ainsi obtenir un arbre phylogénétique. Quelles sont vos conclusions ?
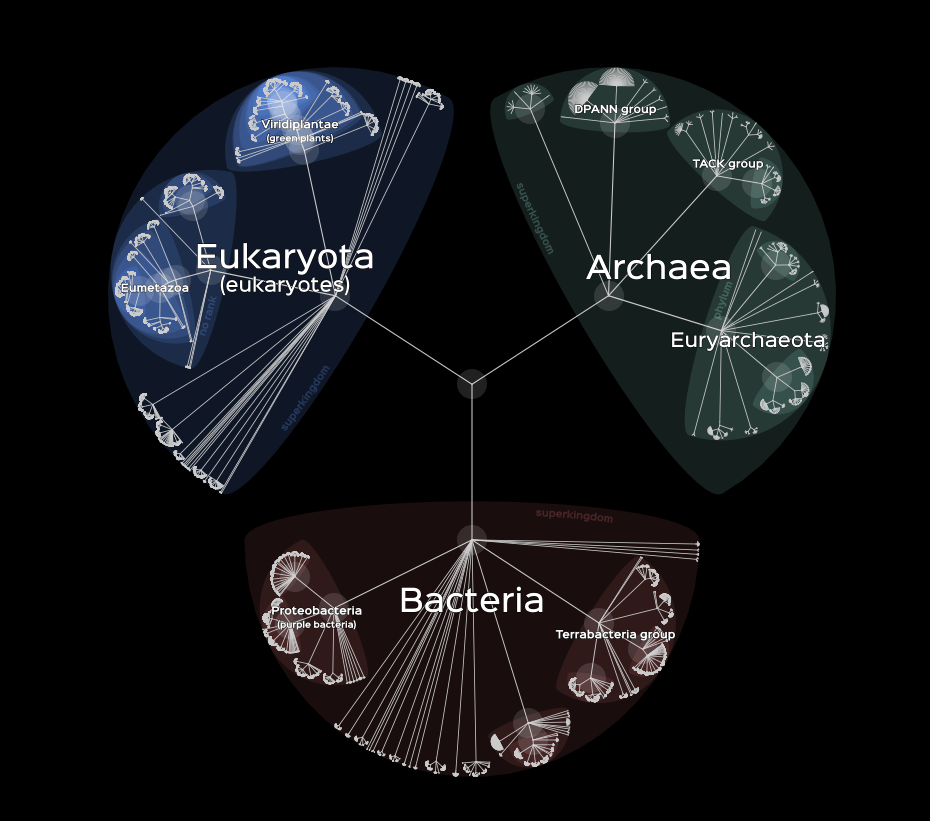
Si vous souhaitez explorer l'arbre de la vie avec l'ensemble des oranismes connus, vous pouvez essayer l'application lifemap:
Conclusion
Nous avons vu ensemble une application de la bioinformatique: la phylogénie moléculaire. La bioinformatique recouvre bien plus de domaines qui nécéssite chacun des compétences et des connaissances particulières. La bioinformatique est devenue maintenant incontournable pour toute analyse moderne de données biologiques notamment par ce que la quantité de données disponibles augmentent tous les jours et qu'il est impossible de pouvoir analyser ses données sans ordinateur. Il existe de nombreuses formations disponibles en bioinformatique à partir de BAC+3 qui forme à différents domaines. Pour finir vous pouvez lire le témoignage d'un bio-informaticien sur son métier et pour en savoir plus sur les formations qui conduisent à devenir bioinformaticien le lien vers la fiche ONISEP